武汉华美生物工程有限公司CUSABIO®品牌商
15 年
手机商铺
- NaN
- 0
- 0
- 2
- 2
推荐产品
公司新闻/正文
技术|制备抗体时抗原的选择
1434 人阅读发布时间:2021-04-15 14:34
一天之计在于晨,一年之计在于春。那么制备一个抗体,抗原的设计和表达系统的选择则至关重要,可以毫不夸张的说,设计出一个好的抗原对于制备一个高质量抗体来说就是成功了一大半。
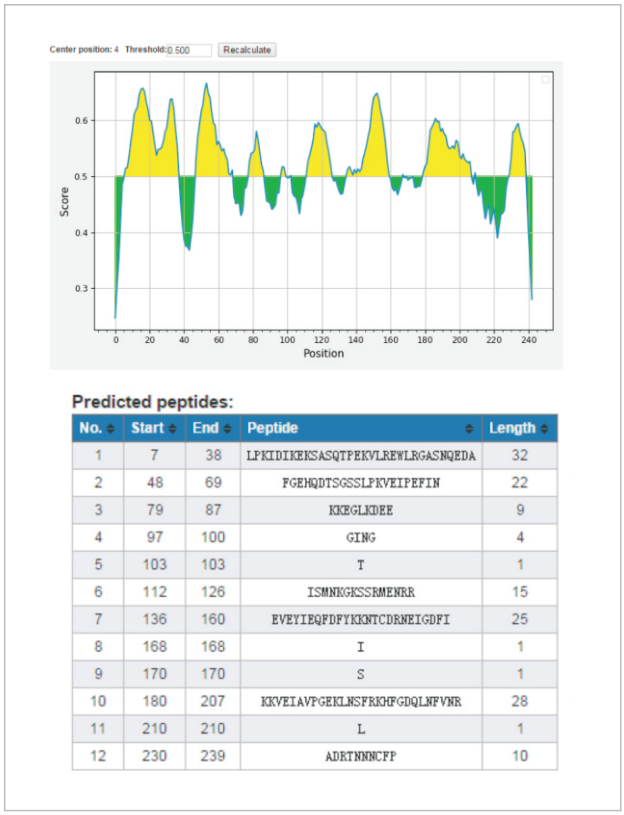
不同的蛋白类型,选择的方法需要多样性。大部分情况下,蛋白C端的亲水性、表面可及性和柔性都相对N端较好。蛋白的二级结构也是预测B细胞表位重要参数之一,β转角为凸出结构,多出现在蛋白质抗原表面,有利于与抗体结合,较可能成为抗原表位。而α螺旋和β折叠结构规则不易变形,较难结合抗体,一般不作为抗原表位。含有5个以上的氨基酸残基的转角又常称为环(loop)。以往的研究表明,蛋白表面的loop区可能为功能性抗体的识别位点,特异性好,可及性强。
Uniprot数据库和CBS服务器(http://www.cbs.dtu.dk/)预测信号肽、跨膜、表位等结构;EX-PASY服务器(http://www.expasy.org/tools)上的GOR4、HNN、SOPMA、nnPredict等方法。亲水性、柔韧性、表面可能性、抗原表位预测、α螺旋和β折叠可以应用DNAstar软件预测,同时可以结合Uniprot数据中的蛋白结构分析。MOE软件也可以预测蛋白的结构,同时MOE软件可以从多个角度分析蛋白的结构特性。
线性表位的预测方法: B细胞表位的预测方法主要集中于线性表位,大量的预测B细胞表位的算法都是基于蛋白质序列。这些算法包括:蛋白质的亲水性算法、可及性算法、蛋白质可塑性算法、蛋白质二级结构预测算法、蛋白质抗原性算法。这些方法的代表软件有PEOPLE、PREDITOP、BEPITOPE、Bcepred、ABCpred、BepiPred、APP等。
构象表位的预测方法:绝大多数B细胞表位预测方法都是基于蛋白质的一级或二级结构的,但这些方法只能用来预测由连续的氨基酸残基构成的线性表位,而基于蛋白质的三级结构来预测构象表位的方法比较少,这是因为各种抗原的构象表位可获得的数据要远远少于线性表位。
基于蛋白质三级结构来预测构象表位的方法CEP:这是第一个以抗原蛋白的三级结构PDB文件作为输入条件,以构象性表位预测为主要目的的网上免费服务软件。由于抗原抗体之间的相互作用属于蛋白质与蛋白质之间相互作用中的一种,因此,可以参这些方法来预测B细胞表位。
分子对接:主要用来研究分子间的相互作用与识别,进而预测复合物结构。常用的分子对接软件有ZDOCK、DOT、DOCK、ClusPro等。
通过对抗原的结构和B细胞表位分析预测后,然后再结合抗体的使用要求,进行选择适当的区间以及表达系统进行抗原表达纯化。
如果是正对与IF、FC、细胞、抗体结合等实验的话,我们推荐的蛋白是全长的或者成熟的跨膜和非跨膜区间,表达系统主要选择无细胞、哺乳、酵母这类具有蛋白活性或者能表达出全长的表达系统进行表达蛋白,对应的抗体类型主要选择单抗和基因工程抗体。
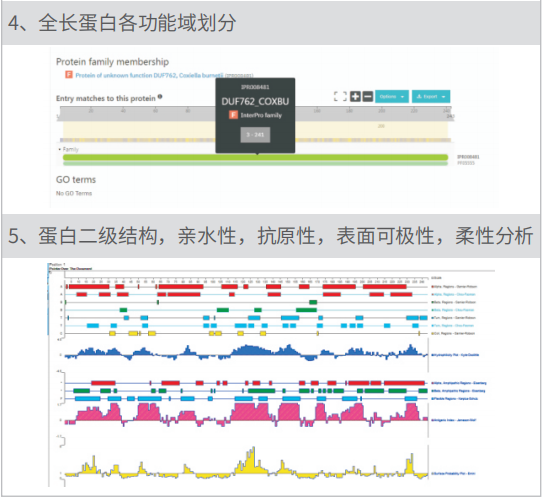
https://psort.hgc.jp/form2.html 亚细胞定位分析
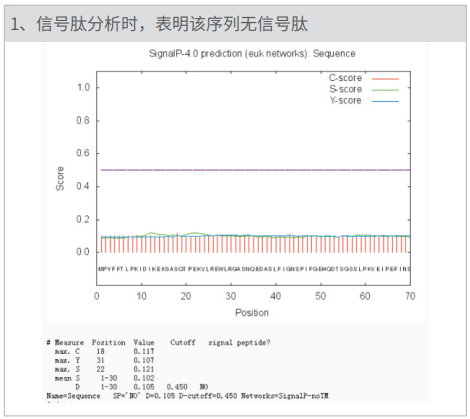
http://www.cbs.dtu.dk/services/SignalP-4.0/ 信号肽分析
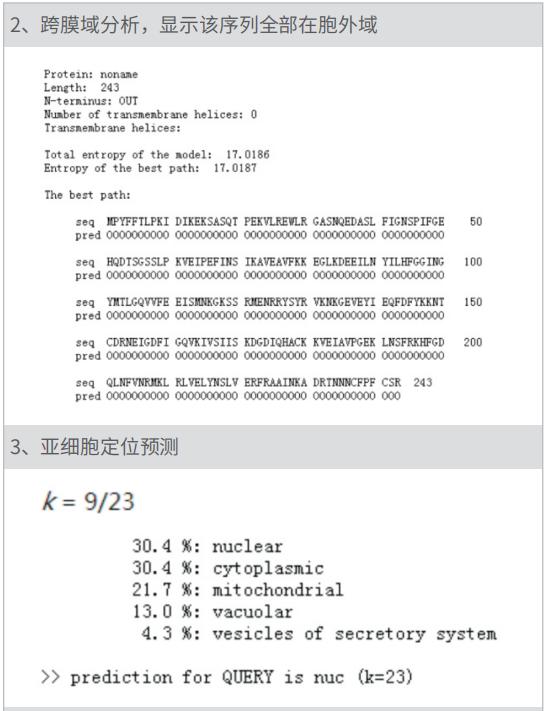
http://www.enzim.hu/hmmtop/html/adv_submit.html 跨膜分析
https://ihg.gsf.de/ihg/mitoprot.html 线粒体转运肽分析
http://nls-mapper.iab.keio.ac.jp/cgi-bin/NLS_Mapper_form.cgi 核定位分析
http://www.proteinatlas.org/ 表达预测(人种属)
https://bgee.org/?page=gene 表达丰度预测
http://old.protein.bio.unipd.it/espritz/ 结构无序性分析
https://swissmodel.expasy.org/ 三级结构预测
http://www.cbs.dtu.dk/services/NetNGlyc/ 糖基化修饰预测分析
https://www.ebi.ac.uk/interpro/ 功能预测分析




